perl 的字符串确实非常厉害,正则就是最完备的没有之一。julia 的正则也是用的 PCRE 的库。
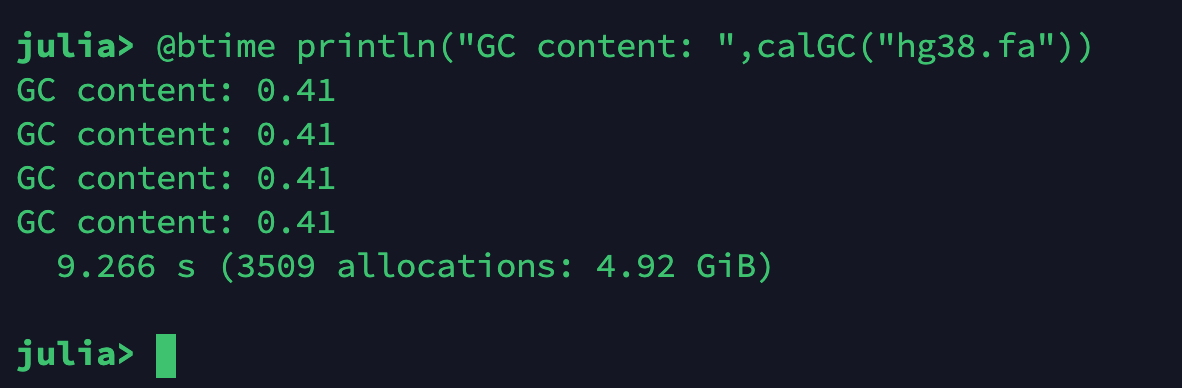
各位,事实证明julia只是在科学计算上具有速度优势,字符串处理只能是普通速度,我使用了经过优化的BioSequence库来处理生物序列,将计算时间争取到了11秒的时间,以下是修改后的代码
using BioSequences
using FASTX
function lineGC(seq)
GCnumber=count(x->(x==DNA_G||x==DNA_C),seq)
lineNum=length(seq)-count(isambiguous,seq)
GCnumber,lineNum
end
function calGC(fs)
GCnumber=zero(Int)
lineNum=zero(Int)
reader=open(FASTA.Reader,fs)
for record in reader
GC,all=lineGC(FASTA.sequence(record))
GCnumber+=GC
lineNum+=all
end
round(GCnumber/lineNum;digits=3)
end
println("GC content: ",calGC(ARGS[1]))
@btime的结果能贴出来吗 ![]()
这个实现可以达到6s
julia> function countChars(io::IO; eol::AbstractChar='\n')
a = Vector{UInt8}(undef, 8192)
nb = 0; arr = zeros(Int, 128)
flag = true
while !eof(io)
nb = readbytes!(io, a)
@inbounds @simd for i=1:nb
(a[i] == UInt8('>')) && (flag = false)
(a[i] == UInt8('\n')) && (flag = true)
(flag) && (arr[a[i]] += 1)
end
end
arr
end
julia> countChars(f::AbstractString; eol::AbstractChar = '\n') = open(io->countChars(io, eol = eol), f)
julia> @time res = countChars(fs);
5.941596 seconds (76.71 k allocations: 4.289 MiB)
julia> res['C' |> Int] + res['c' |> Int]
623727342
这个确实很快,但是我总觉得很多人学juliah就是拿来即用的程度,而你这版本,我感觉非常非常像C语言或者C++
Julia对字符串的速度本身就不是很好,毕竟要做到支持非ASCII码会出现一些性能牺牲。简单的工作还是用UInt8数组好一些。
println去掉吧,直接@btime calGC(...);
记得加分号
原来的代码里面有个闭包,而这是已知的类型不稳定。。。
function calGC(fs)
GCnumber=zero(Int)
lineNum=zero(Int)
open(fs,"r") do IOstream
for line in eachline(IOstream)
if startswith(line,">")
continue
else
GC,all=lineGC(line)
GCnumber+=GC
lineNum+=all
end
end
end
round(GCnumber/lineNum;digits=3)
end
具体说来,do 后面的函数引用了位于calGC中的两个局部变量,而导致这两个变量被Box
所以正确的做法是把变量移动到do函数里面。
是的,我本来也是想学习一下Julia的,但是,在字符串处理方面,它一点优势都没用,为了速度写的复杂了,还不如go语言,尽管我根本不知道怎样把Julia写的像go一样快,但是我可以轻而易举地把它写到和python 一样慢 唉,学习都没动力了
说起来Python3到Python2的升级把字符串的表示从Bytes的原生(原始)表示改成Unicode效率也下降30%来着。。只能说本来在Python2里用字符串表示的问题在Python3的对应中就不应该用字符串来表示。直接对应的考虑效率自然降低。
作为生物信息工作者,个人认为julia在生信分析上相比R缺少太多包了,大火的CRISPR,RNAseq,单细胞的都没有对应的处理程序。相比Python则很多C库缺失例如htslib相关的pysam, pyfaidx等快速处理测序和比对数据的库函数。如果自己写算法的话还要时刻注意算法的优化,最后写着写着更像静态语言了。希望julia的生态能够赶超python和R, 但是目前还不行
使用numpy矩阵的方式寻找GC含量,按照字节Byte的方式查找
时间需要2分钟,是比perl和julia慢不少
import sys
from pyfaidx import Fasta
import numpy as np
def lineGC(seq):
gc_number = np.where((seq==b'G')|(seq==b'C')|(seq==b'g')|(seq==b'c'))[0].shape[0]
n_number = np.where((seq==b'N')|(seq==b'n'))[0].shape[0]
allnumber = seq.shape[0] - n_number
return (gc_number,allnumber)
def calGC(fs):
GC = 0
all = 0
hg38 = Fasta(fs)
for record in hg38:
seq = np.asarray(record)
gc_number,all_number=lineGC(seq)
GC = GC + gc_number
all = all + all_number
return (GC, all)
if __name__ == "__main__":
gcNum, allNum = calGC(sys.argv[1])
print("GC content: {:.3f}".format(gcNum/allNum))
字符串不能随机访问,把字符串当作数组来处理本来就是违反了字符串的接口(尤其是去索引这个字符串)。所有正确实现的字符串的处理,必然都和Perl和Julia一样慢。只不过很多人以为这世界上所有的字符串都是ASCII格式的,但是这并不对。
因此正确的做法就是把数据用readbytes读入到Vector{UInt8}中按照一个个Byte处理。
你要实在是想要用字符串的话,就用Base.CodeUnits包装字符串,然后字符串就像一个数组了。。。
顺便再吐嘈一下这个帖子,题主提供的用来Benchmark数据差不多有1个GB大,对于想帮助你的人来说下载是很麻烦的事情(譬如在我的网络环境下下载要差不多4-5个小时),应当提供一个较小的数据集(1~10MB)(你甚至可以随机生成一个也没有问题)。
简单说,提问者应该提供一个MWE(Minimal Working Example最小可行实例)+有代表性的数据集。
我强烈建议社区管理员开一个“提问须知”置顶帖,并把这一点写进去,同样的还有要求每个人显示BenchmarkTools的测试结果和自己电脑的versioninfo()。。。
- 非常抱歉由于我糟糕的提问让你感到困扰,社区管理早就有提问须知在置顶中,是我个人并没有遵守该条约,因为个人也是初学,对于
Benchmarktools知之甚少,而官方文档中的性能建议我读完后并不能完全理解其中的意思,因此再此提问 julia无论是文档还是深有研究的前人,对于小白用户非常不友好。- 我使用了
transcode将字符串转化为UInt8的数组,性能并没有显著提升,既然字符串索引会影响性能,julia提供多种影响性能的方法或函数在官方文档中,是否是一种误导?官方对该语言的宣传让我以为该语言既有python的优雅又可以具有高性能,然后事实上两者依旧无法兼得。
-
“既然字符串索引会影响性能,julia提供多种影响性能的方法或函数在官方文档中,是否是一种误导?”——然而正确的字符串就是这么个性能,所以不是字符串错了,是你用错数据结构了。无论在哪个语言,UTF的字符串就是这样。。。如果它很快,那就是它的实现错了。除非你认为不做安全检查的字符串是好的(因此在索引的时候可能返回各种non-sence的值)。Julia文档在这里做的不好的一点是:它理应挂一个横幅在字符串一节下,警告人们不要直接把字符串当作字节数组处理。。。
-
用transcode是肯定不正确的,因为你要的是字符串的底层表示,而transcode会新生成一个数组,其内容是原来字符串的转码。因此真的要把字符串看成字节数组,就要用Base.CodeUnits(s),s是一个字符串,这个方法返回一个可索引对象。
-
“官方对该语言的宣传让我以为该语言既有python的优雅又可以具有高性能,然后事实上两者依旧无法兼得。“,然而你自己的例子就证伪了这一点,在用了Biosequece,jl以后你的程序时间只有Perl的1/3,而代码还几乎不变,而你对应的Numpy的Python代码更慢。即使没用Biosequence,在修正了你的类型不稳定以后也减少到了1/2的时间。
而更加重要的是,这个回答下没有人指出这一点:这个程序有两个部分构成,一个IO部分和一个计算部分,然而所有人都在Benchmark整个函数,你这样怎么知道到底是IO部分很慢(考虑到这个文件有整整一个GB大),还是计算部分很慢呢?@ AquaIndigo利用读入字节缓冲数组的方法时间减小到了只有5秒左右(实际上我们还可以进一步优化),从某种方面说明了,可能大部分的时候都花在了读入构造短字符串(以及count遍历短字符串引起的overhead)。。。至于计算的部分,可能还无关紧要。
我说一个计算部分可以用的优化吧,在做多个判断的时候,可以用一个trick减少分支,见知乎的这个回答:C 语言有什么奇技淫巧? - 知乎
x==‘A’ || x==‘B’ 写成 (x-‘A’)|(x-‘B’) == 0
当然这要求我们的字符必须是ASCII编码(也就是要转换成UInt8)的。。。
好的多谢,我再研究研究